前回に引き続き、今回も日本版(1)のデータの前処理編です。
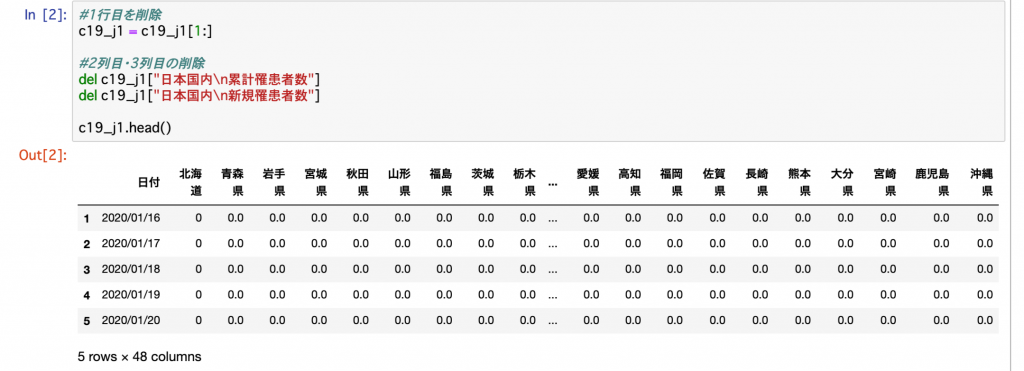
前回はデータフレームc19_j1から不要な行を1つ・列を2つ削除するところまで行いました。
これで日付と都道府県だけのデータになりましたが、これだと今の私にはPythonで分析出来る状態にありません。
その為、もう一手間加える必要があります。
具体的には日付・罹患者数・都道府県の3列になるように処理を加えたいと思います。
しかし、その前に一つ行っておかなければいけないことを忘れていました。
欠損値の確認です。
最後に行って必要な情報も消してしまっては元も子もないので、遅ればせながら欠損値の確認を行おうと思います。
c19_j1.isnull().sum()で欠損値の個数を確認したところ、日付以外は全て15個の欠損値が存在することが分かりました。
さらに
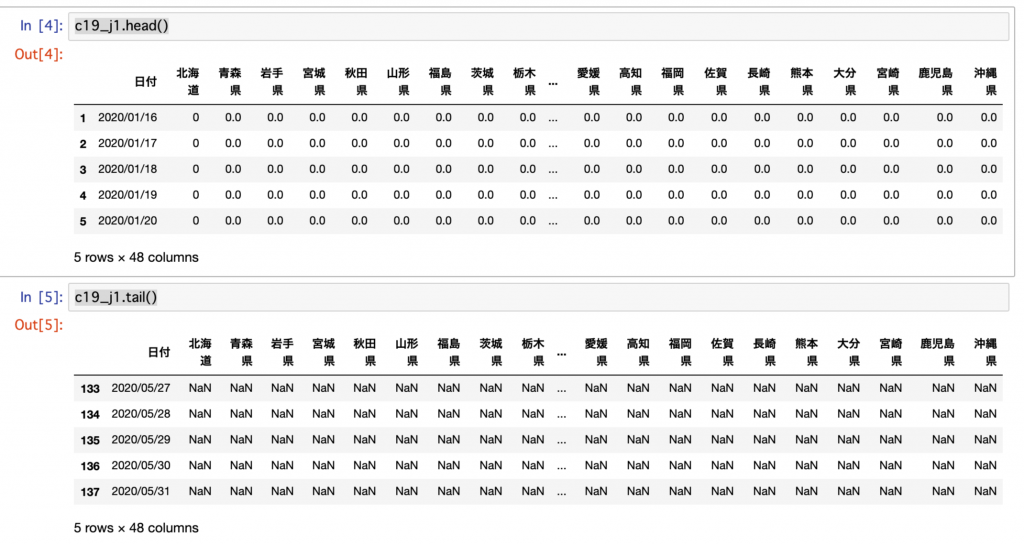
c19_j1.head()
c19_j1.tail()で 、先頭と末尾の内容を確認します。
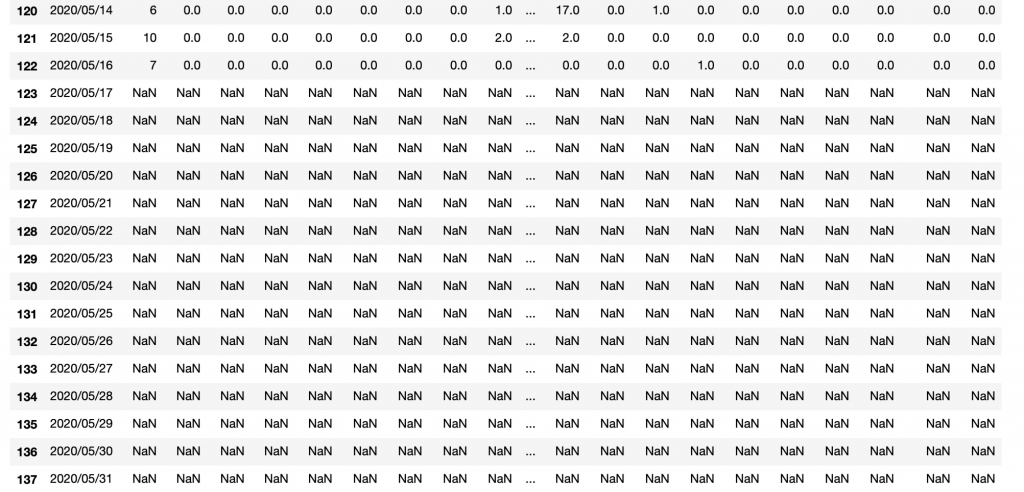
末尾を見ると20200527〜20200531が全てNaNで欠損値になっています。
さらに末尾からの表示範囲を広げて調べると、20200517以降が全て欠損値であることが分かりました。
これは元データであるSIGNATE COVID-19 Case Datasetをダウンロードしたのが2020/05/16だったので、2020/05/17以降がなくて当然。
データとして正しいと判断できます。
欠損値が存在するのは20200517以降。
これなら欠損値をまとめて削除しても問題がなさそうです。
c19_j1 = c19_j1.dropna()
c19_j1.isnull().sum()で、欠損値を削除し、あらためて欠損値の個数を確認します。
15個あった欠損値が0になり、欠損値がなくなったことが確認できました。
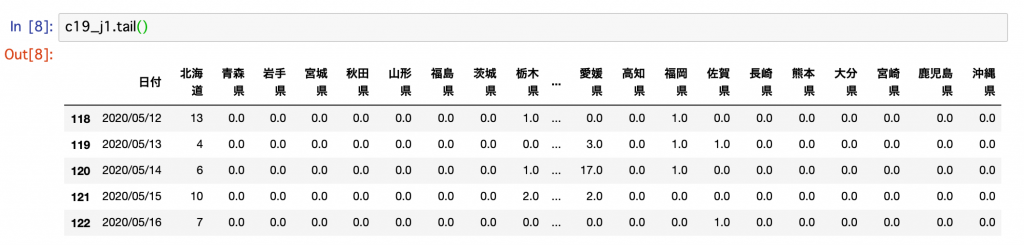
念の為に末尾も確認しておきます。
無事、欠損値のある20200527〜20200531が削除され、20200116〜20200516のデータになっています。
このデータをさらに処理して、次回の作業を進めようと思います。