今回から本格的なプログラムの変更を行いたいと思います。
前回は新たに元データとなる厚生労働省のオープンデータの内容確認と今後の方針確認を行いました。
今回から本格的なプログラムの変更を行いたいと思います。
まずは各データのカラムのリネームを行います。
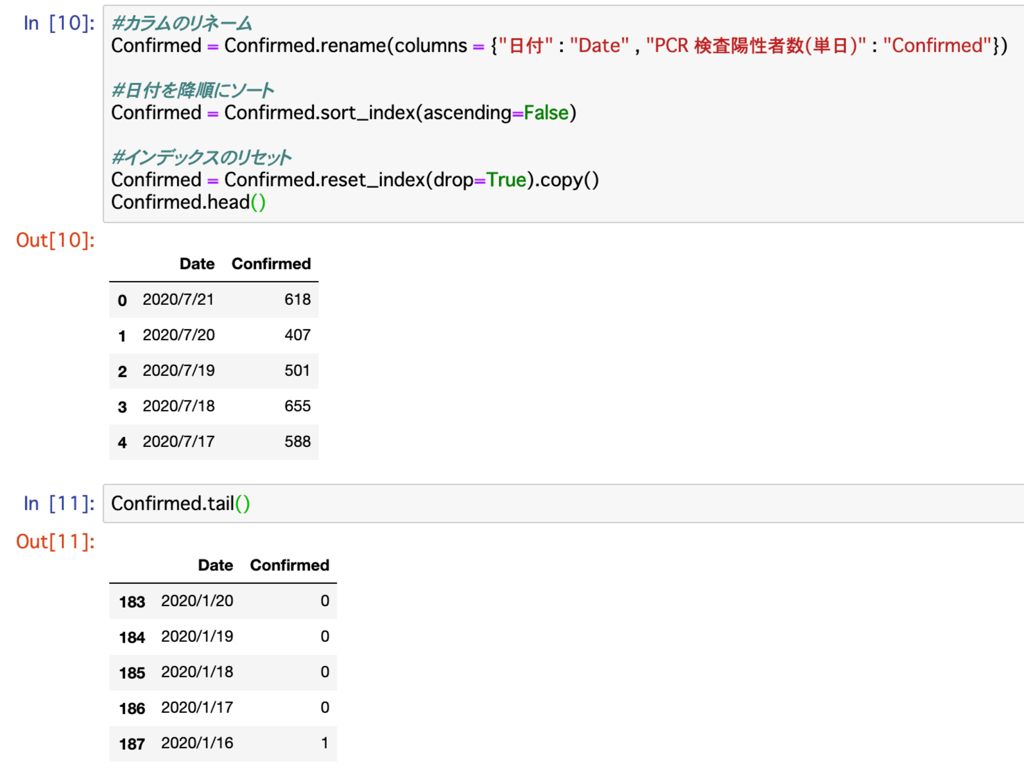
Confirmedは日付をDate、 PCR 検査陽性者数(単日)をConfirmed
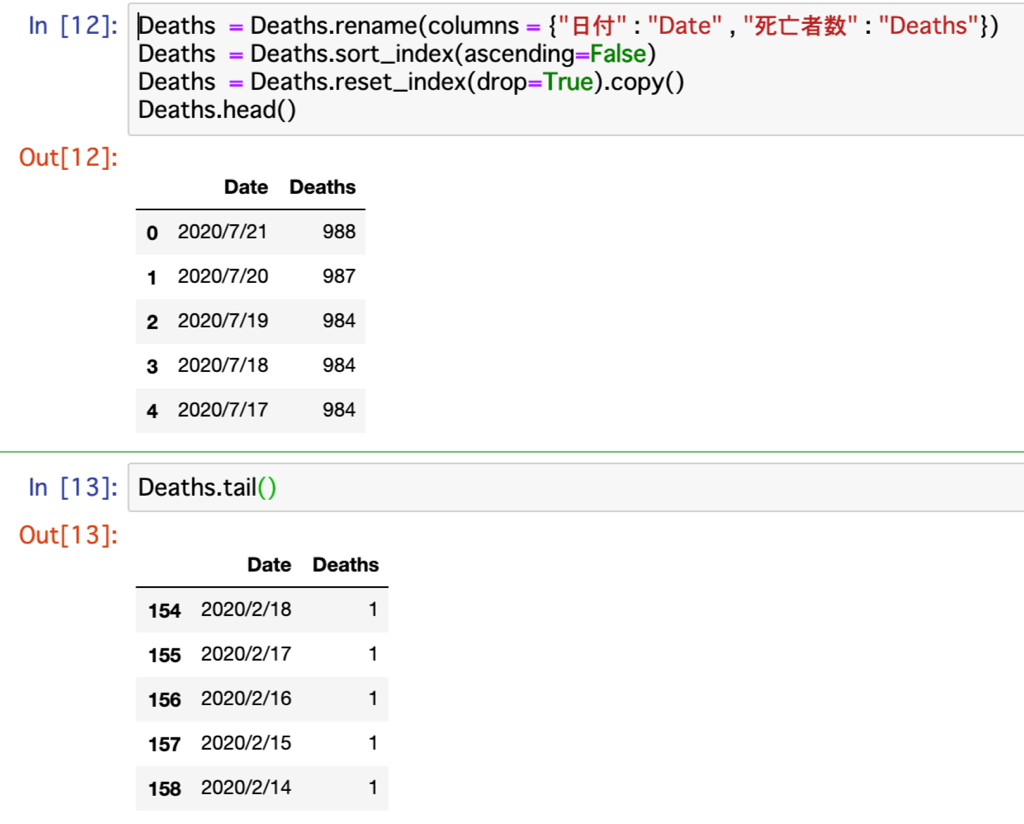
Deathsは日付をDate、 死亡者数をDeaths
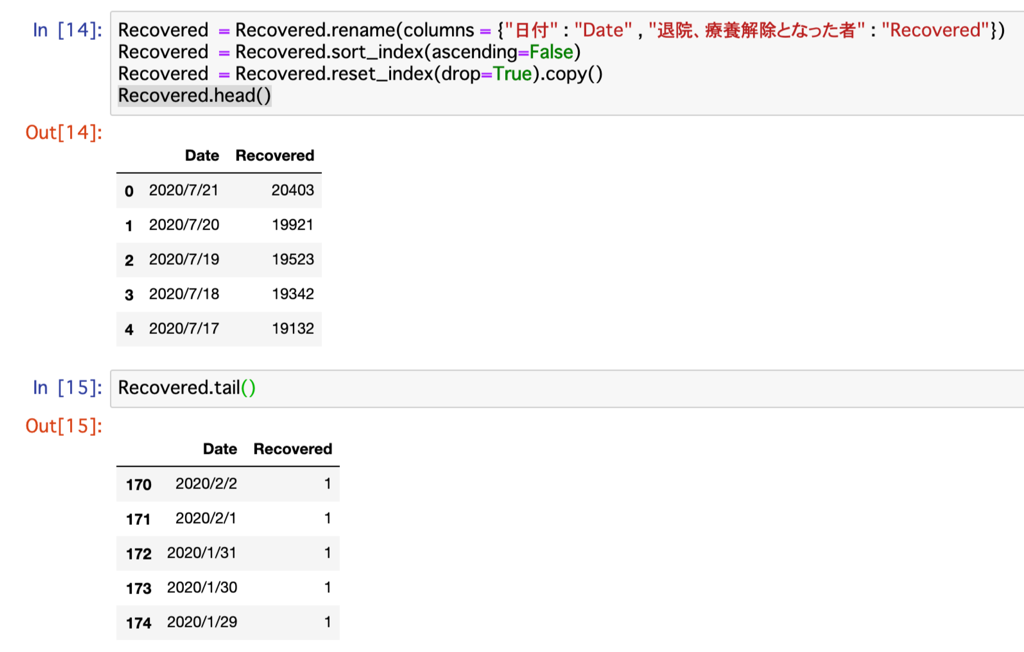
Recoveredは日付をDate、 退院、療養解除となった者をRecovered
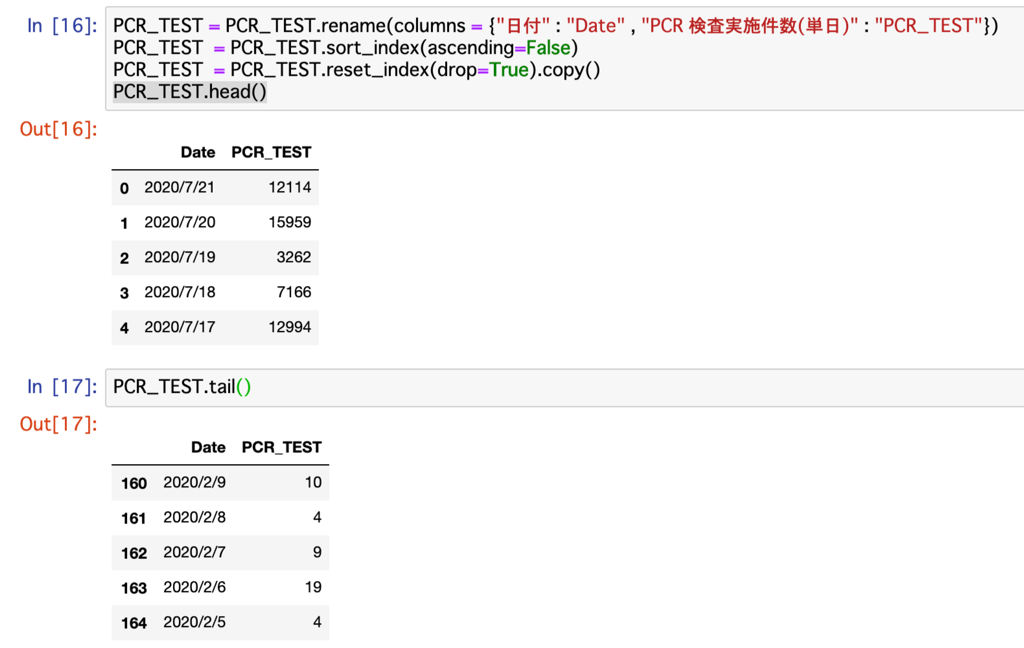
PCR_TESTは日付をDate、 PCR 検査実施件数(単日)をPCR_TEST
にそれぞれ変更します。
同時に、DeathsとRecoveredの日別数字への変換と今後のデータの結合の為に、日付を降順になるようにソートも行います。
まずはConfirmedからテストを兼ねて行います。
こちらが上手くいけば、後は同じ処理を残り3つに適用します。
以下のコードを実行してカラムのリネーム、日付のソート、念の為にインデックスのリセットも行います。
Confirmed = pd.read_csv("./Origina_data/" + "pcr_positive_daily.csv" , encoding = "utf=8")
Confirmed.head()
Confirmed.tail()
Deaths = pd.read_csv("./Origina_data/" + "death_total.csv" , encoding = "utf=8")
Deaths.head()
Deaths.tail()
Recovered = pd.read_csv("./Origina_data/" + "recovery_total.csv" , encoding = "utf=8")
Recovered.head()
Recovered.tail()
PCR_TEST = pd.read_csv("./Origina_data/" + "pcr_tested_daily.csv" , encoding = "utf=8")
PCR_TEST.head()
PCR_TEST.tail()完成したデータを確認するとカラムのリネーム、日付のソート、インデックスのリセットが問題なく行えています。
Deaths・Recovered・PCR_TESTにも同じ処理をそれぞれに行うことにします。
Deaths = Deaths.rename(columns = {"日付" : "Date" , "死亡者数" : "Deaths"})
Deaths = Deaths.sort_index(ascending=False)
Deaths = Deaths.reset_index(drop=True).copy()
Deaths.head()
Deaths.tail()
Recovered = Recovered.rename(columns = {"日付" : "Date" , "退院、療養解除となった者" : "Recovered"})
Recovered = Recovered.sort_index(ascending=False)
Recovered = Recovered.reset_index(drop=True).copy()
Recovered.head()
Recovered.tail()
PCR_TEST = PCR_TEST.rename(columns = {"日付" : "Date" , "PCR 検査実施件数(単日)" : "PCR_TEST"})
PCR_TEST = PCR_TEST.sort_index(ascending=False)
PCR_TEST = PCR_TEST.reset_index(drop=True).copy()
PCR_TEST.head()
PCR_TEST.tail()Deaths・Recovered・PCR_TESTもカラムのリネーム、日付のソート、インデックスのリセットが問題なく行えたことが確認されました。
次にデータフレームの結合を行います。
既に日別数字になっているConfirmedとPCR_TEST、これから期間累計数字を日別数字にDeathsとRecoveredとをそれぞれ横に結合します
keyには列Dateを使用し、merge()メソッドで結合を行います。
ConfirmedとPCR_TESTをマージしてデータセットc19j2_day1
DeathsとRecoveredをマージしてデータセットc19j2_total
を作成します。
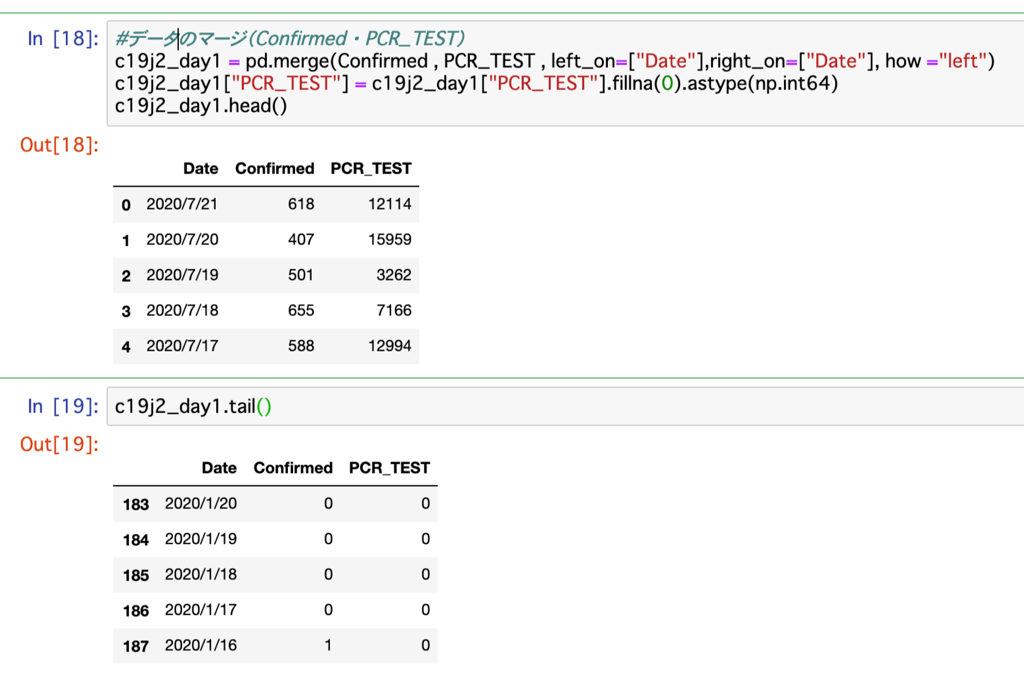
#データのマージ(Confirmed ・ PCR_TEST)
c19j2_day1 = pd.merge(Confirmed , PCR_TEST , left_on=["Date"],right_on=["Date"], how ="left")
c19j2_day1["PCR_TEST"] = c19j2_day1["PCR_TEST"].fillna(0).astype(np.int64)
c19j2_day1.head()
c19j2_day1.tail()
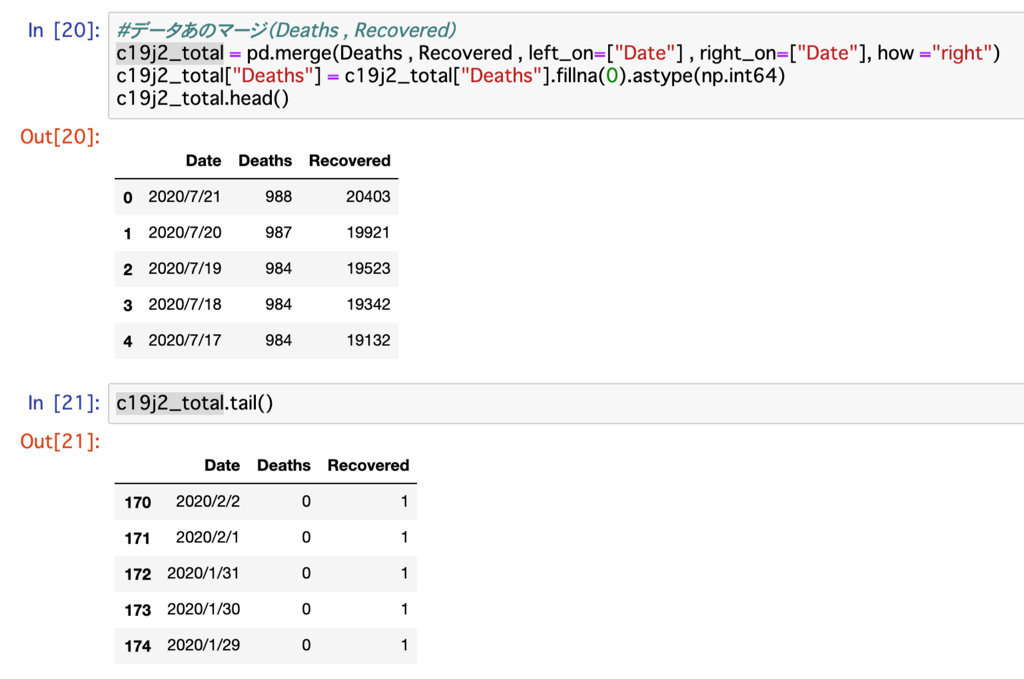
#データあのマージ(Deaths , Recovered)
c19j2_total = pd.merge(Deaths , Recovered , left_on=["Date"] , right_on=["Date"], how ="right")
c19j2_total["Deaths"] = c19j2_total["Deaths"].fillna(0).astype(np.int64)
c19j2_total.head()
c19j2_total.tail()マージする場合に注意したのは、データの長さの違いです。
データのどちらを優先するかでデータの長さが違ってしまう為、それを念頭に置いて指示をする必要があります。
データセットc19j2_day1の場合、Confirmedが1/16 , PCR_TESTが2/5なので、日付の長いConfirmedをhow =”left”で、
データセットc19j2_totalの場合、Deathsが2/14 , Recoveredが1/29なので、日付の長いRecoveredをhow =”right”で、
それぞれ指定します。
完成したデータを確認するとデータセットc19j2_day1は1/16、データセットc19j2_totalは1/29と、それぞれデータの長い方のデータの長さでマージされています。
また、長さの合わないデータの方は、既存データは整数から小数に(なぜか?)変換、足りない分が欠損値として補間されます。
その為、その列に関しては、
.astype(np.int64)でキャストして小数を整数に戻し、
欠損値は.fillna(0)を使って0で補完
する処理も同時に行っていることを追記しておきます。
後は通期累計数字になっているデータセットc19j2_totalを日別数字に変換し、そのデータとデータセットc19j2_day1を結合すれば完成です。
しかし、まだもう少し時間がかかりそうなので、それらは次回に行おうと思います。