苦労の末、可視化に必要なプログラムc19W_df_ver1.0の完成に至った世界版でしたが、残念ながら期間累計数字の為、直近の数字の変化が直感的に見えず、新たなフェーズに入った現在のニーズにはそぐわない結果となってしまいました。
その為、この以下プログラムに手を加え、日別の累計数字になるように変更を行うことにします。
import pandas as pd
import numpy as np
#Recovered基本データセットの読み込み
c19w_df = pd.read_csv("time_series_covid19_recovered_global.csv" , encoding = "utf=8")
#不要列の削除
del c19w_df["Province/State"]
del c19w_df["Lat"]
del c19w_df["Long"]
#同じ名前が複数ある国の確認
c19w_df["Country/Region"].value_counts().head(10)
#value_countsのデータから、列Country/Regionに同じ国名が複数ある国の選択
scl = c19w_df["Country/Region"].value_counts().head(10)
scl = scl.index[0:6]
scl
#列Country/Regionに同じ国名が複数ある国の抽出・csv書き込み・再読み込み
for sc in scl:
sc_df = c19w_df.loc[c19w_df["Country/Region"] == sc]
sc_df = sc_df.groupby(["Country/Region"]).sum()
sc_df.to_csv(""+ sc + "_df.csv", encoding = "utf=8")
sc_df2 = pd.read_csv(""+ sc + "_df.csv", encoding = "utf=8")
China_df2 = pd.read_csv("China_df.csv", encoding = "utf=8")
France_df2 = pd.read_csv("France_df.csv", encoding = "utf=8")
UnitedKingdom_df2 = pd.read_csv("United Kingdom_df.csv", encoding = "utf=8")
Australia_df2 = pd.read_csv("Australia_df.csv", encoding = "utf=8")
Netherlands_df2 = pd.read_csv("Netherlands_df.csv", encoding = "utf=8")
Denmark_df2 = pd.read_csv("Denmark_df.csv", encoding = "utf=8")
#結合予定6カ国の行をオリジナルデータからの削除
c19w_df = c19w_df[c19w_df["Country/Region"] != "China"]
c19w_df = c19w_df[c19w_df["Country/Region"] != "United Kingdom"]
c19w_df = c19w_df[c19w_df["Country/Region"] != "France"]
c19w_df = c19w_df[c19w_df["Country/Region"] != "Australia"]
c19w_df = c19w_df[c19w_df["Country/Region"] != "Netherlands"]
c19w_df = c19w_df[c19w_df["Country/Region"] != "Denmark"]
#結合予定6カ国のオリジナルデータからの削除の確認
c19w_df["Country/Region"].value_counts().head(10)
#c19w_dfのcsvファイルへの書き出し・再読み込み
c19w_df.to_csv("c19w_df.csv", encoding = "utf=8")
c19w_df2 = pd.read_csv("c19w_df.csv", encoding = "utf=8", index_col=0)
#合計データとオリジナルデータの結合
c19w_df3 = pd.concat([China_df2, UnitedKingdom_df2, France_df2, Australia_df2, Netherlands_df2 , Denmark_df2 , c19w_df2], axis=0)
#行と列の変換
c19w_df4 = c19w_df3.T
#c19w_df4のcsvファイルへの書き出し・再読み込み
c19w_df4.to_csv("c19w_df4.csv", encoding = "utf=8", header = None)
c19w_df5 = pd.read_csv("c19w_df4.csv", encoding = "utf=8")
#Date・Recovered・Countryの3列のカラムに変換処理
#作成したデータフレームを格納する空のリストを作成
lo_df = []
#カラムの国名をfor分で回す
for i in c19w_df5.columns[1:] :
#日付と該当する国の感染者数の二列のデータを作成
x = c19w_df5[["Country/Region",i]].copy()
#カラム「Countryを作成し、該当する都道府県名を入力
x["Country"] = i
#二列目の国名iを「Confirmed」に変更
x = x.rename(columns={ i :"Recovered"})
#1列目のカラムを「Country/Region」から「Date」に変更
x = x.rename(columns={ "Country/Region" :"Date"})
#変数xをデータフレームlo_dfに格納
lo_df.append(x)
X = lo_df[0]
#年月日を格納する空のリストを作成
ymd_l = []
y_l = []
m_l = []
d_l = []
for i in range(len(X["Date"])) :
y = X["Date"][i].split('/')[2]
y = "20" + y
y_l.append(y)
for i in range(len(X["Date"])) :
m = X["Date"][i].split('/')[0]
if len(m) == 1:
m = "0" + m
m_l.append(m)
else:
m_l.append(m)
for i in range(len(X["Date"])) :
d = X["Date"][i].split('/')[1]
if len(d) == 1:
d = "0" + d
d_l.append(d)
else:
d_l.append(d)
nums = len(m_l)
for num in range(nums):
ymd = y_l[num] + m_l[num] + d_l[num]
#print(len(ymd))
#print(ymd)
ymd_l.append(ymd)
#日付のフォーマットを変換
#年月日を格納する空のリストを作成
ymd_l = []
y_l = []
m_l = []
d_l = []
for i in range(len(X["Date"])) :
y = X["Date"][i].split('/')[2]
y = "20" + y
y_l.append(y)
for i in range(len(X["Date"])) :
m = X["Date"][i].split('/')[0]
if len(m) == 1:
m = "0" + m
m_l.append(m)
else:
m_l.append(m)
for i in range(len(X["Date"])) :
d = X["Date"][i].split('/')[1]
if len(d) == 1:
d = "0" + d
d_l.append(d)
else:
d_l.append(d)
nums = len(m_l)
for num in range(nums):
ymd = y_l[num] + m_l[num] + d_l[num]
#print(len(ymd))
#print(ymd)
ymd_l.append(ymd)
#バラバラの188国分のデータセットを1つに結合
c19w_Recovered = pd.concat(lo_df , sort=False)
c19w_Recovered.head()
#データフレームc19w_Recoveredに新しい列Date_new行を追加
c19w_Recovered["Date_new"] = pd.DataFrame(ymd_l)
c19w_Recovered.head()
#不要になった列Dateの削除
del c19w_Recovered["Date"]
#カラムの並び替え
c19w_Recovered = c19w_Recovered[["Date_new","Country","Recovered"]]
#カラムのリネーム
c19w_Recovered= c19w_Recovered.rename(columns={"Date_new" : "Date"})
#csvファイルへの書き込み
c19w_Recovered.to_csv("c19w_Recovered.csv",index =None , encoding = "utf=8")
#csvファイルからの読み込み
c19w_Recovered = pd.read_csv("c19w_Recovered.csv" , encoding = "utf=8")
c19w_Recovered.head()以前から、何回か試みてはいたものの、うまく進まない状況が続いていました。
しかし、このほど、同様の問題に直面していた日本版(2)の方が無事にプログラムのバージョンアップに成功。
世界版も同じ要領でこちらもバージョンアップを行いたいと思います。
基本的に行う処理は同じ。
期間累計数字なので、当日から前日の数日をマイナスし、日別の数字を算出するという作業を行います。
日本版(2)では日本一国のみに処理をおこなえば良かったのですが、世界版では188カ国に同様の処理をおこなわなければなりません。
しかし、for分を使えば、それほど苦にはならない問題のはずです。
次回から実際に、プログラムの追加・修正をいきたいと思います。
期間累計数字を可視化する為のデータセットは既に作成済みの為、どこかのどこかの段階で、当日から前日の数日をマイナスする作業という、日別累計数字に変換すれば良いわけです。
どの段階で行うかが問題となりますが、それはプログラムの処理の流れを追えば自然と決まりました。
上記のように、indexに日付・カラムに188カ国が並んだデータセットを作成した後に、それぞれの国に対して日別数字に変換する処理を行い、しかる後に、再びVer1の処理に戻ると言う流れになります。
ところがこれが思ったよりも手間取りました。
日本版(2)では日本一国を処理すれば良かったのですが、
前回、ようやく可視化に必要なデータセットが完成しました。
今回はいよいよ可視化を行いたいと思います。
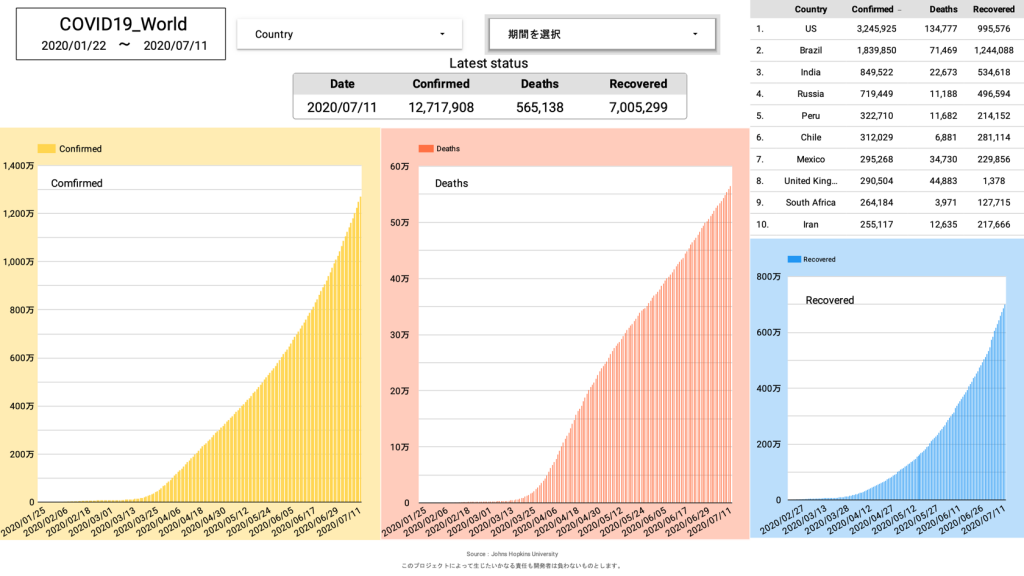
まず、個人的に実現したかったのは全世界の感染者数・死亡者数・回復者数がひと目で分かるグラフ。
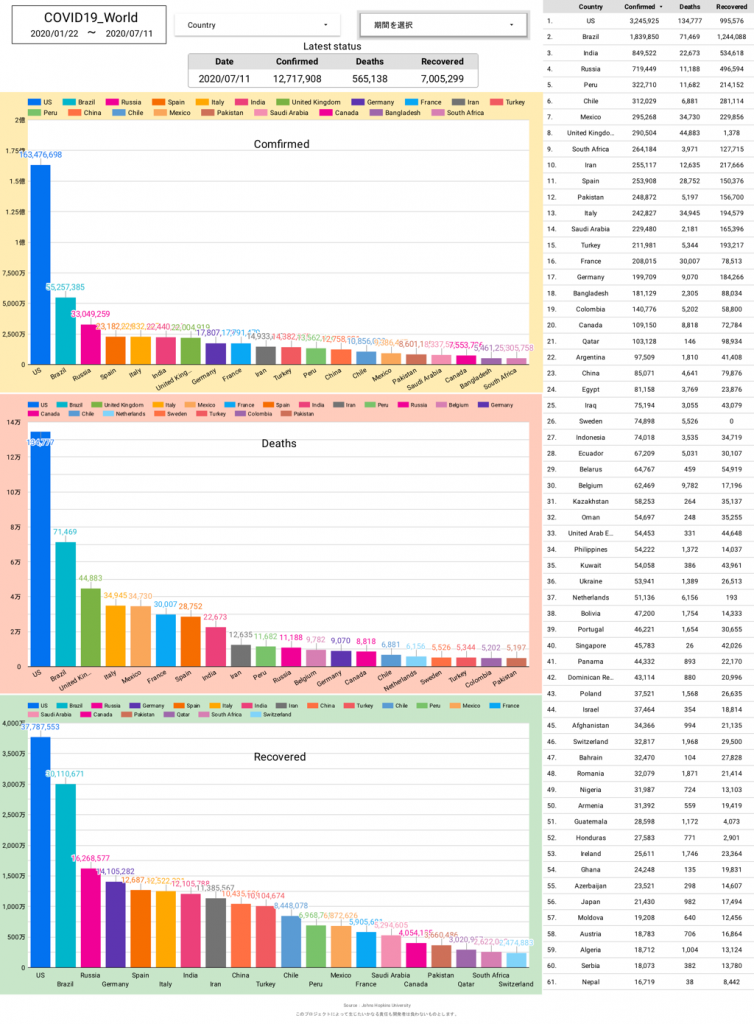
次に、188カ国ある内のトップ10〜20迄の国の感染者数・死亡者数・回復者数がひと目で分かるグラフ。
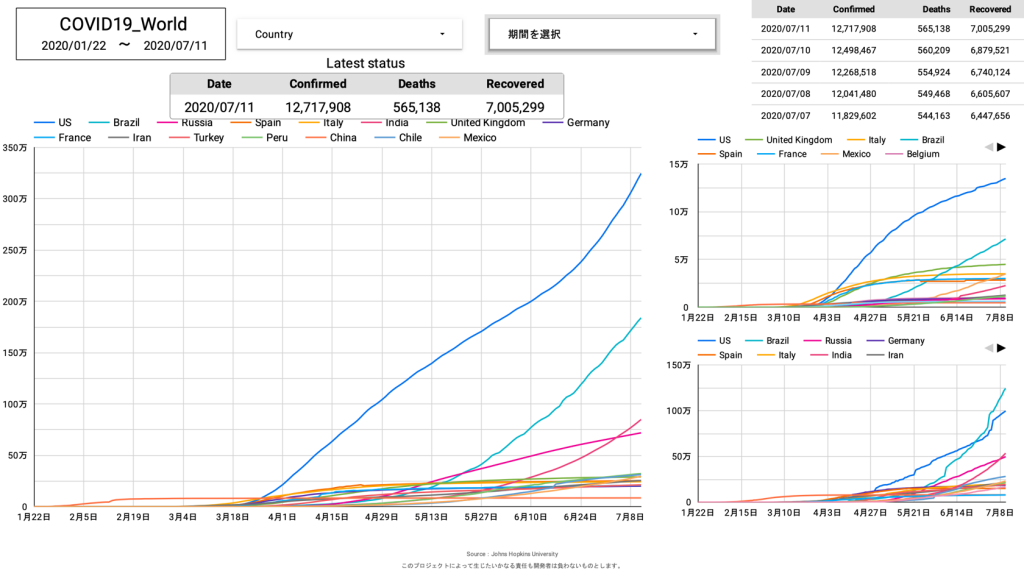
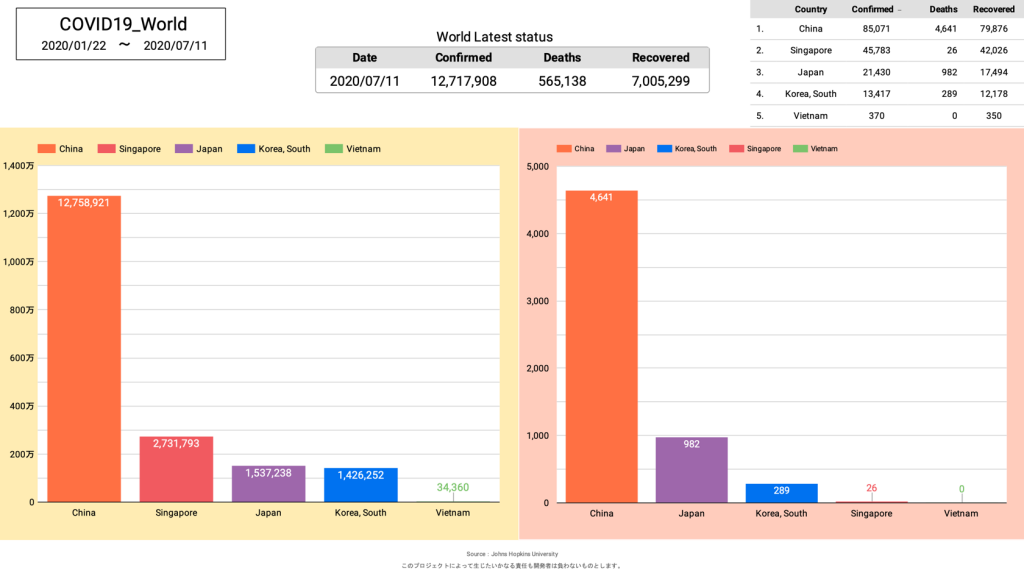
さらに、自分が住む日本と他の国々との比較ができるグラフ。
上記三種類が当面必要としているグラフとなります。
しかし、日本版の時もそうでしたが、感染者数・死亡者数・回復者数のグラフを限られた表示内に効率的に配置するのは難しく、今後も試行錯誤が必要です。
WEBなどを参考にしながら、今後も随時バージョンアップを図りたいと思います。
ともあれ、世界版もこれで一段落付きました。
日本版(2)の時もそうですが、直近の変化を直感的に理解するには、期間全体の累計だけでなく、やはり日毎の数字が欲しい。
理論的には現在のデータセットを変更することで、日毎の数字を算出できるはずなので、日本版(2)とも併せて、今後早期に実現させたいと思います。