Part4に引き続きデータの前処理編となります。
Part4では日付データの整形を行い、年・月・日とばらばらの列だった日付を、年月日表示にまとめ、新しい列Dateを追加し、そこへ格納するところまでを行いました。
今回は追加した列の並び替えとリネームを行い、最後に出来上がったデータをCSVファイルに書き込むところまでを行おうと思います。
まずは列の並び替えから。
前回作成した列Dateが問題なく作成できているかを、元データの列であるyear・month・dateと比較します。
しかし、列Dateとyear・month・dateが離れていて見にくい。
その為、現在右端にある列Dateを、見やすいようにyear・month・dateのすぐ右隣に移動します。
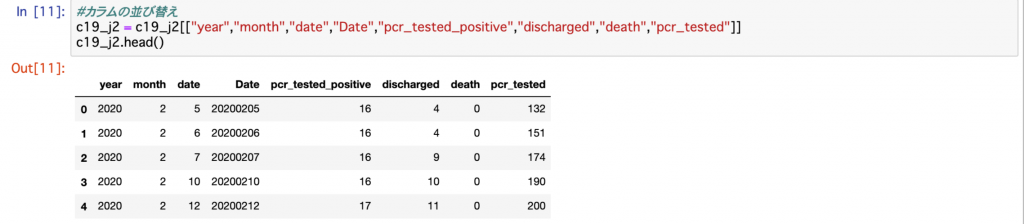
c19_j2 = c19_j2[["year","month","date","Date","pcr_tested_positive","discharged"
,"death","pcr_tested"]]
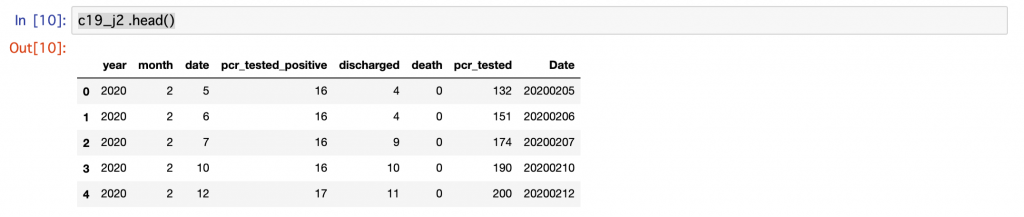
c19_j2.head()上記コードを実行して表示された結果が以下となります。
日付を確認すると2020年2月5・6・7日と連続し、それから10日に飛び、さらに一日飛んで12日と特徴的な並びですが、year・month・dateとDateは問題なく同じ表示になっています。
末尾も確認しておこうと思いますが、同時にリネームを行いたいと思います。
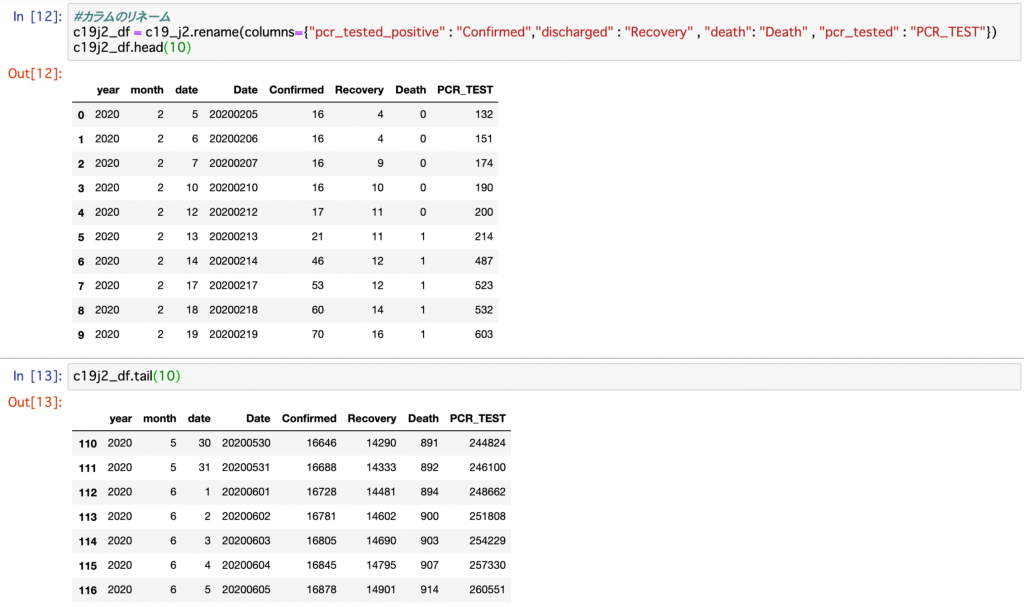
c19j2_df = c19_j2.rename(columns={"pcr_tested_positive" : "Confirmed"
,"discharged" : "Recovery" , "death": "Death" , "pcr_tested" : "PCR_TEST"})
c19j2_df.head(10)
c19j2_df.tail(10)上記コードを実行して表示された結果が以下。
今回は追加した列Dateの変換が正確に行われているかを確認するために、先頭と末尾をそれぞれ10行ずつ表示しました。
PCR検査陽性のpcr_tested_positiveを感染者のConfirmed、退院のdischargedを回復者のRecovery、deathはDeath、pcr_testedはPCR_TESTにそれぞれリネームされています。
日付も先頭・末尾共に問題なく変換されていることが確認されました。
これでyear・month・dateは不要です。
以下コードで削除します。
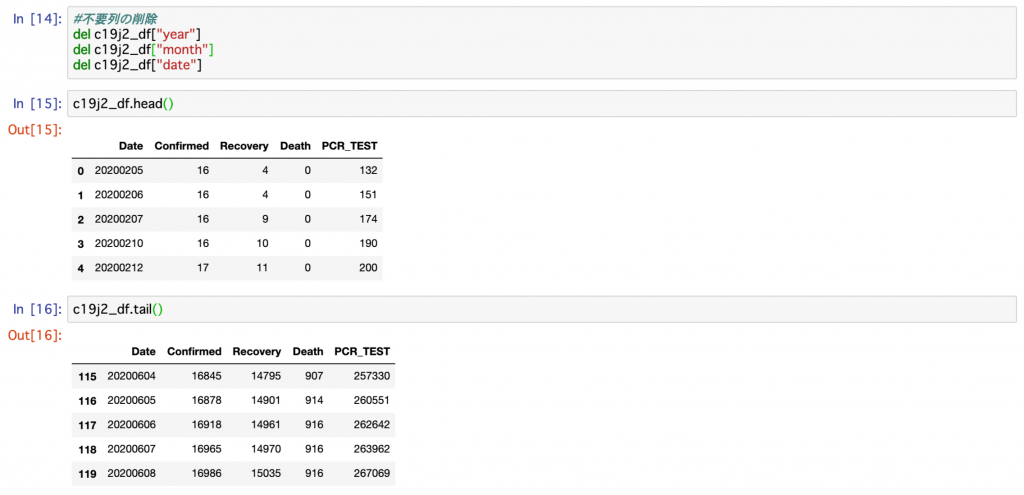
#不要列の削除
del c19j2_df["year"]
del c19j2_df["month"]
del c19j2_df["date"]これでデータを可視化するのに必要なデータが完成しました。
最後に欠損値の有無と内容の確認を行っておきます。
それぞれ以下コードを実行します。
c19j2_df.isnull().sum()
c19j2_df.info()欠損値はなく、最初と同じ120行のデータを保持できています。
後は以下コードを実行してCSVファイルに書き出しをして前処理は完了となります。
出来上がったプログラムはc19j2_df_ver1.0と呼称しようと思います。
ようやく可視化・分析にも取り組めるようになりました。
しかし、このプログラグムが最新のデータに対して問題なく動作するかどうかが分かりません。
内容が変更されていると正しく動作しない可能性があり、必要ならコードを修正する必要があります。
分析・可視化へと進む前に、次回は最新のデータでc19j2_df_ver1.0の動作検証を行おうと思います。